oncology
ΤruSight Oncology
500 panel
Η πλέον εξατομικευμένη
προσέγγιση στον καρκίνο σήμερα
Το ΤruSight Oncology 500 της Illumina, αποτελεί την πιο ολοκληρωμένη και αποτελεσματική υπηρεσία ανάλυσης ενός πλήρους γονιδιωματικού προφίλ όγκων μετά από αλληλούχιση νέας γενιάς (NGS) 523 γνωστών γονιδίων που σχετίζονται με ΟΛΟΥΣ τους τύπους συμπαγών όγκων και κάποιων αιματολογικών νεοπλασιών. Πρόκειται για μια ολιστική προσέγγιση, από τον έλεγχο του δείγματος έως τις προτεινόμενες επιλογές σε στοχευμένες θεραπείες, ανοσοθεραπείες και κλινικές δοκιμές, που επιτρέπει στους ιατρούς να παρέχουν την καλύτερη δυνατή θεραπευτική προσέγγιση και φροντίδα στους ασθενείς τους.
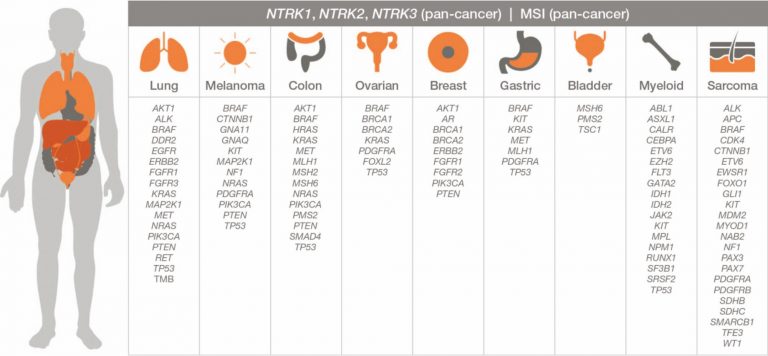
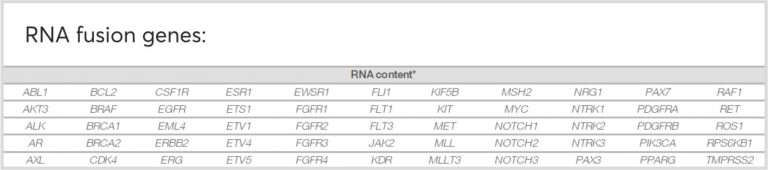
Συγκεκριμένα, το TruSight Oncology 500 ανιχνεύει όλους τους τύπους παραλλαγών που μπορεί να σχετίζονται με καρκινογένεση, όπως μονονουκλεοτιδικές παραλλαγές (SNVs), μικρού μεγέθους ελλείψεις και προσθήκες (indels), παραλλαγές ωρίμανσης του RNA (splice variants), υβριδικά μετάγραφα (RNA fusions), ενώ επίσης είναι η πλέον αξιόπιστη μέθοδος ανίχνευσης του φορτίου μεταλλάξεων του όγκου (ΤΜΒ, Tumor Mutational Burden) και της Μικροδορυφορικής Αστάθειας (ΜSI, Microsatellite Instability):
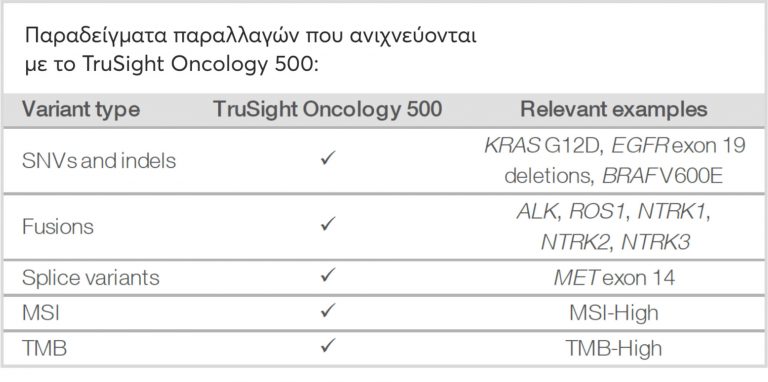
Αυτές οι πληροφορίες παρουσιάζονται σε αναλυτική, εξατομικευμένη έκθεση αποτελεσμάτων, από την επικυρωμένη και διαπιστευμένη πλατφόρμα κλινικής ανάλυσης δεδομένων PerianDx (www.periandx.com), που εξηγεί τη βιολογική σημασία κάθε μεταβολής, με βιβλιογραφικές αναφορές, καθοριστικές για τη λήψη αποφάσεων με στόχο τη βέλτιστη διαχείριση της νόσου.
Συγκριτικά με άλλες παρόμοιες λύσεις, τα πλεονεκτήματα της ανάλυσης με TruSight Oncology 500 είναι τα εξής:
- To TruSight Oncology 500 καλύπτει τον μεγαλύτερο αριθμό γονιδίων (523) από όλα τα άλλα τεστ τα οποία έχουν επιλεχθεί με βάση τις κατευθυντήριες οδηγίες του NCCN και καλύπτουν το 100% των βιοδεικτών για τους αναφερόμενους τύπους καρκίνου. • Οι περιοχές που καλύπτονται είναι συγκριτικά περισσότερες, δηλαδή 1,94 εκατομμύρια βάσεις και άρα παρέχει ακριβέστερη εκτίμηση του ΤΜΒ.
- Χρειάζεται το λιγότερο αρχικό δείγμα ιστού ή DNA, δηλαδή 5 τομές παραφίνης (10μm) ή 40ng DNA.
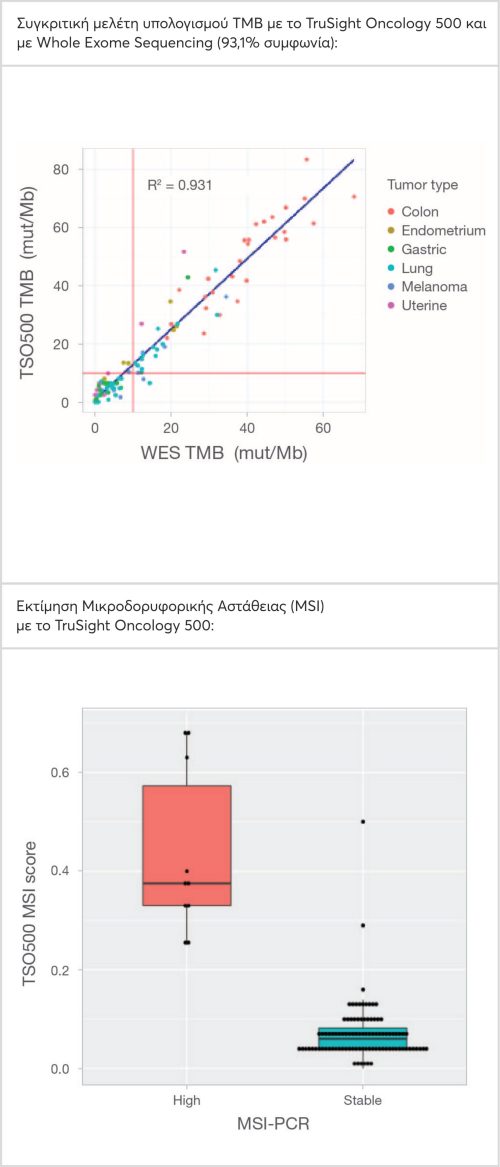
- Υπολογίζει ταυτόχρονα τη μικροδορυφορική αστάθεια (MSI) με ιδιαίτερα μεγάλη ακρίβεια, ενώ περιλαμβάνει επίσης και αναδυόμενους βιοδείκτες στον καρκίνο, όπως: POLD1, POLE1 και HLA.
- Είναι βασισμένο στην τεχνολογία NGS με ανιχνευτές τύπου UMI (Unique Molecular Identifiers) για μεγαλύτερη ακρίβεια και ευαισθησία, εκμηδενίζοντας τα επιμέρους τεχνικά σφάλματα.
- Η αναλυτική ευαισθησία του τεστ είναι > 96% (για όλους τους τύπους των παραλλαγών σε συχνότητα παραλλαγμένου αλληλομόρφου 5%).
- Η αναλυτική ειδικότητα της μεθόδου είναι > 99.99%.
- Έχει τη μεγαλύτερη συμφωνία στα αποτελέσματα του ΤΜΒ score συγκριτικά με Whole Exome Sequencing, δηλαδή 93,1%.
- Είναι η μόνη διαθέσιμη λύση στην αγορά που ανιχνεύει χιμαιρικά γονίδια (fusion genes), πληροφορία πολύ μεγάλης σημασίας για την επιλογή φαρμάκων σε κάποιες περιπτώσεις (πχ Ca Πνεύμονα).
- Πραγματοποιείται σε διαπιστευμένο εργαστήριο στην Ελλάδα με χρόνο απάντησης 2-3 εβδομάδες, ενώ ο αρχικός έλεγχος ποιότητας του δείγματος (και πιθανή αναζήτηση επανάληψης) ολοκληρώνεται εντός 2-3 ημερών.
- Τα πρωτογενή αποτελέσματα της αλληλούχισης αναλύονται από επικυρωμένη πλατφόρμα με διεθνή αναγνώριση που εγγυάται, εκτός άλλων, την αποφυγή ψευδώς θετικών (low confidence) αποτελεσμάτων, καθώς και την απομάκρυνση των κληρονομούμενων παραλλαγών (germline filtering) μέσω σύγκρισης με πληθυσμιακές βάσεις δεδομένων.
- Το εναπομείναν δείγμα του ασθενούς (DNA), καθώς και τα δεδομένα της ανάλυσης είναι πάντα στη διάθεση του ασθενούς και του παραπέμποντος ιατρού για μελλοντικές επανεκτιμήσεις και αναλύσεις.
Επιλεγμένη Βιβλιογραφία
- Stransky N, Cerami E, Schalm S, Kim JL, Lengauer C. The landscape of kinase fusions in cancer. Nat Commun. 2014;5:4846. doi:10.1038/ncomms5846.
- Boland GM, Piha-Paul SA, Subbiah V, et al. Clinical next generation sequencing to identify actionable aberrations in a phase I program. Oncotarget. 2015;6(24):20099-20110.
- Massard C, Michiels S, Ferté C, et al. High-Throughput Genomics and Clinical Outcome in Hard-to-Treat Advanced Cancers: Results of the MOSCATO 01 Trial. Cancer Discov. 2017;7(6):586-595.
- Harris MH, DuBois SG, Glade Bender JL, et al. Multicenter Feasibility Study of Tumor Molecular Profiling to Inform Therapeutic Decisions in Advanced Pediatric Solid Tumors: The Individualized Cancer Therapy (iCat) Study. JAMA Oncol. 2016. doi: 10.1001/jamaoncol.2015.5689.
- Parsons DW, Roy A, Yang Y, et al. Diagnostic Yield of Clinical Tumor and Germline Whole-Exome Sequencing for Children With Solid Tumors. JAMA Oncol. 2016. doi: 10.1001/jamaoncol.2015.5699.
- Zehir A, Benayed R, Shah RH, et al. Mutational landscape of metastatic cancer revealed from prospective clinical sequencing of 10,000 patients. Nat Med. 2017;23(6):703-713.
- Illumina (2017) TruSight Oncology UMI Reagents. (www.illumina.com/content/dam/illumina marketing/documents/products/datasheets/trusight-oncology-umi-reagents-datasheet-1000000050425.pdf).
- NCCN Guidelines. www.nccn.org/professionals/physician_gls/default.aspx. Accessed December 13, 2018.
- Tray N, Weber JS, Adams S. Predictive Biomarkers for Checkpoint Immunotherapy: Current Status and Challenges for Clinical Application. Cancer Immunol Res. 2018;6(10):1122-1128.
- Buchhalter I, Rempel E, Endris V, et al. Size Matters: Dissecting Key Parameters for Panel-Based Tumor Mutational Burden (TMB) Analysis. Int J Cancer. 2018. doi: 10.1002/ijc.31878.
- Chalmers ZR, Connelly CF, Fabrizio D, et al. Analysis of 100,000 human cancer genomes reveals the landscape of tumor mutational burden. Genome Med. 2017;9(1):34. doi: 10.1186/s13073-017-0424-2.
- Illumina (2018) Analysis of TMB and MSI Status with TruSight Oncology 500. (emea.illumina.com/content/dam/illumina-marketing/documents/products/appnotes/trusight-oncology-500-tmb-analysis-1170-2018-009.pdf).
- Vanderwalde A, Spetzler D, Xiao N, Gatalica Z, Marshall J. Microsatellite instability status determined by next-generation sequencing and compared with PD-L1 and tumor mutational burden in 11,348 patients. Cancer Med. 2018;7(3):746-756.
- Kautto EA, Bonneville R, Miya J, et al. Performance evaluation for rapid detection of pan-cancer microsatellite instability with MANTIS. Oncotarget. 2017;8(5):7452-7463.
